matplotlibのimshowでヒートマップを高速描画する際の、値の記入や軸ラベルの設定方法
前回、matplotlibでヒートマップを高速描画する簡単な方法について書いた。
spcx8.hatenablog.com
上記の方法は、普通のヒートマップでは描画速度が著しく落ちるようなデータの多いケースに有効。裏を返すと、データの少ない場合(例えば行10x列10とか)は、seabornのヒートマップでも速度は変わらない。そして細かい設定をしたい場合はのseabornの方が何かと便利だったりする。
色で表している値を数字でも表示したかったり、各行・各列のラベルを軸に表示したい場合、seabornは簡単にできる。
参考:Seaborn でヒートマップを作成する – Python でデータサイエンス
でもmatplotlibで押し通すことも可能で、以下のコードがその例。
結果:
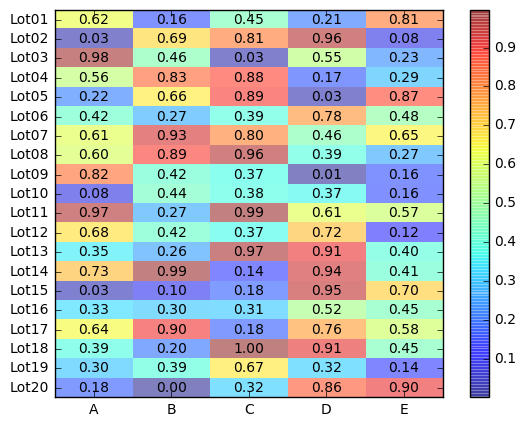
matplotlibのimshowをヒートマップ の表示に使い、その値の数字表示と軸ラベルの調整もmatplotlibのみで行なっている。
もう他のライブラリを覚えるのも面倒だどか、matplotlibを使いこなすことが苦じゃない場合にどうぞ。
matplotlibのimshowでヒートマップを高速描画する際の縦横比(アスペクト比) 調整
前回、matplotlibでヒートマップを高速描画する簡単な方法について書いた。
spcx8.hatenablog.com
これの欠点は、グラフの縦横比が固定されるので、plt.figure(figsize=(*,*)) だけでは調節できないこと。ただ、それもplt.imshow()の引数を調整すれば問題ない。
まずは、縦横比調整無しの場合のコード。
結果:
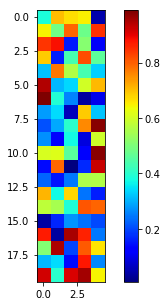
ここでは、図のサイズについてfigsize=(7,5)とやや横長で指定しているが、縦長のグラフになってしまっている。(imshowで20x5ピクセルの絵として描画しているので当たり前の結果)
次に縦横比を調節したコード。
結果:
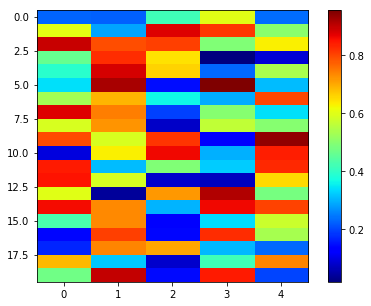
plt.imshow()の引数aspectで描画する絵の縦横比を調整している。ここでは、aspect=0.25 とすることで、横に比べて縦の長さを0.25倍にしている。
matplotlibでヒートマップを高速描画する簡単な方法
Pythonでのヒートマップの描き方を調べると、だいたい以下の2つの方法が出てくる。
- seabornライブラリのheatmap関数を使う方法
Seaborn でヒートマップを作成する – Python でデータサイエンス
- matplotlibライブラリのpcolor関数を使う方法
Python + matplotlib によるヒートマップ
これらの真っ当なヒートマップは、データの大きいと描画に時間がかかるのが欠点。特にseabornは時間がかかる。seabornは使い慣れたら便利そうだしグラフの見た目も綺麗なだけに残念。
速くしたい時は以下のコードがおすすめ。
結果:

画像描画のimshowを使い、引数を interpolation='nearest' とすることで、ヒートマップと同じ表示ができる。この引数を設定しないと、データ間を補間して表示してしまう(データ間の境界がはっきりしなくなる)。
100万画素のデータ(行数1000 x 列数1000)のデータの場合で計算時間を計測してみたら、以下のように圧倒的な速さだった。
- seaborn.heatmapを使った場合 : 35 sec
- matplotlib.pcolorを使った場合 : 6 sec
- matplotlib.imshowを使った場合 : 0.5 sec
欠点はグラフの縦横比が固定されるので、plt.figure(figsize=(*,*)) だけでは調節できないことくらい。これの対処法は以下の記事を参照。
ちなみに各行列のラベル表記はしない前提。というか行数・列数多くてラベルをつけることが無理なような大きいデータを使うから関係ないですね。
マトリクス形式 (配列) から スプレッド形式 (表) に変換するときも numpy.meshgrid が役立つ
マトリクス形式(配列)のデータをスプレッド形式(表)に変換したい場合がある。
例えば下のような配列を、、
[[0.36363082, 0.00332102, 0.14533355],
[0.11039035, 0.95780551, 0.48281694],
[0.44771602, 0.94823649, 0.36095112]]
↓ こんなふうな表にしたいという意味。
|
row
|
col
|
val
|
|
|---|---|---|---|
| 0 | 0 | 0 | 0.190805 |
| 1 | 0 | 1 | 0.599887 |
| 2 | 0 | 2 | 0.209723 |
| 3 | 1 | 0 | 0.105745 |
| 4 | 1 | 1 | 0.219456 |
| 5 | 1 | 2 | 0.198315 |
| 6 | 2 | 0 | 0.671719 |
| 7 | 2 | 1 | 0.537687 |
| 8 | 2 | 2 | 0.012490 |
工夫なしに書くとこんなコードになる。
これにはもっと高速にできる方法があって、npのmeshgridという関数を使う。これを使うとfor構文が不要になり、コードもシンプル。
ちなみにこれ、前回書いた内容とやってる事はほとんど同じで、使い道がちがうだけでした。
2次元配列の値を行番号や列番号に依存させたい場合、meshgrid()を使って高速化
2次元配列を作るときに、中身の値を行番号や列番号に依存させたい場合がある。
つまり↓のような関係が成り立つようにしたい。
2次元配列の値 = 関数( 行番号, 列番号 )
普通に書くとこうなる。
ここでは配列の値 = 行番号 + 列番号 となるようにしている。
このコードで作ったmatを一応可視化すると、
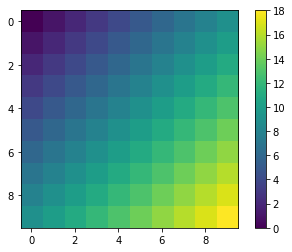
右下に行くほど値が大きくなっている。
そしてもっといい方法がこちら。
numpy にはmeshgridという便利な関数が用意されていて、forを使わずに書くことができる。
結果は同じなので割愛。
ちなみに、meshgridを使うと処理速度も改善できる。というかmeshgridを使うのはこれが目的だったりする。
nrows, ncols ともに 10,000の場合で計測した結果、
- 前述のコード(for2段階) 31.6 sec
- 後述のコード(meshgrid) 1.8 sec
とその差は明らか。自分でfor構文を書くのは極力やめた方がいいらしい。
pandas / DataFrame の中身の文字列を一括で分割して別の列として定義する
pandasで、特定の列の(中身の)文字列を分割して使いたいときがある。
例えばこんな表があったとして、
|
NumName
|
|
|---|---|
| 0 | a001_name1 |
| 1 | a002_name2 |
| 2 | a003_name3 |
↓ こんな風にしたい
|
NumName
|
num
|
name
|
|
|---|---|---|---|
| 0 | a001_name1 | a001 | name1 |
| 1 | a002_name2 | a002 | name2 |
| 2 | a003_name3 | a003 | name3 |
この処理をするのに、以前はこんなもっさりしたコードを書いていた。
分割したい列の中身を取り出して、1行ずつ分割処理するやり方だ。
でも下のようにたった2行で書けることがわかった。
for を使わないので処理速度も上がるかもしれない。(pandasが裏で効率よくやってくれているはず)
pandas公式ページ参照:
https://pandas.pydata.org/pandas-docs/stable/generated/pandas.Series.str.split.html
ちなみ1行目にある .str が便利なやつで、これを使うと文字列の処理を列単位で一括でやってくれる。文字列操作 replace() もご覧の通り。
|
NumName
|
num
|
name
|
NumPlace
|
|
|---|---|---|---|---|
| 0 | a001_name1 | a001 | name1 | a001_place1 |
| 1 | a002_name2 | a002 | name2 | a002_place2 |
| 2 | a003_name3 | a003 | name3 | a003_place3 |
pandasのおかげで本当に仕事がはかどる。
ダイアログボックスでファイル名を指定したい(Windows/Macでコードが違う)
データを取り込む時にはファイル名を指定する。
pd.read_csv()でも np.loadtxt()でもそう。
でファイル名を指定するのにコードをいちいち書き換えたくないので、
ダイアログボックスを使う方法を調べた。
調べたらtkinterというライブラリを使うみたいだが、これがPython2系とPython3系でコードが色々と違うみたい。Python3系向けにここでメモ。
OSが違うとコードの書き方が変わるので注意。
windowsの場合
引数filetypesでダイアログボックスに表示される拡張子を制限指定できる。ここではcsv,txtに絞ってるけど、すべて表示したければ,
filetypes=[('all files','*.*')]
とすればOK。
Macの場合
macOSとwindowsではfiletypesの書き方が違うようで、上のコードではファイルが指定できなかった。こう書けば大丈夫だった。
ただ、windowsの時のように全てのファイルが選択できるようにする方法はわかりません。
最後に、どちらの場合も一緒にimportしているosは現在のディレクトリを取得するのに使っているだけなので不要なら引数の部分と一緒に削除してOK。